O solo carrega nutrientes e microrganismos, sendo que esses nutrientes são essenciais para os seres vivos (principalmente para as plantas, que dependem dos nutrientes e do solo para o crescimento e desenvolvimento delas) enquanto alguns micróbios possuem habilidade de transferir compostos (como carbono, nitrogênio, fósforo, potássio e entre outras substâncias químicas e biológicas) para a superfície terrestre por meio de atividades metabólicas fazendo partes desses ciclos biogeoquímicos, realizar manutenção da fertilidade do solo, controle de doenças e pragas da agricultura, e propriedades de biorremediação do meio ambiente.
A diversidade de micróbios no solo está relacionado com o estudo de todos os aspectos biológicos do microrganismo (bactéria, archaea, fungo, vírus, protozoário, nematódeo, artrópode, anelídeo, e parasita) que existem nesse ambiente, e o desenvolvimento de novas aplicações para caracterização de comunidades microbiológicas e imagens de ambientes do solo tem beneficiado a microbiologia por fornecer diversas técnicas de detecção e localização de microrganismos, além de detalhar as funções bioquímicas desses organismos com o ambiente os quais habitam.
Métodos de identificação de microrganismos no solo (Bioinformática)
Na área da bioinformática, é utilizável os equipamentos e/ou dispositivos que possam otimizam os dados e processar os métodos de sequenciamento de maneira mais simples do que os métodos mais tradicionais (como por exemplo, dependência de cultura). E as práticas bioinformatas pode-se conectar junto com as áreas de biologia molecular e celular, genética, ciência da computação e TI, estatística e matemática; e os tópicos abaixo são diferentes práticas mais mencionadas para identificação microbiana, sendo que a maioria deles utilizam-se métodos de sequenciamento de novas gerações (NGS) como complemento:
● Metabarcoding: utiliza sequências específicas para identificar quais espécies estão presentes na amostra e como elas estão relacionadas filogeneticamente (utiliza-se a aplicação adequada com NGS); ○ NGS: tecnologia de sequenciamento de DNA ou RNA que realiza leitura de milhões de fragmentos de maneira simultânea, tendo uma análise em larga escala e rápido (mas apenas individual).
● Metagenômica: prática de sequenciamento de genomas inteiros de comunidades microbianas isoladas, e quais tipos estão presentes e como é a dinâmica entre eles;
● Biosensing (definição de IUPAC): “um dispositivo que utiliza reações bioquímicas específicas mediadas pelas enzimas isoladas, sistemas imunológicos, tecidos, organelas, ou células inteiras para detectar compostos químicos geralmente por meio de sinais elétricos, térmicos ou ópticos”.
○ NGS: tecnologia de sequenciamento de DNA ou RNA que realiza leitura de milhões de fragmentos de maneira simultânea, tendo uma análise em larga escala e rápido (mas apenas individual).
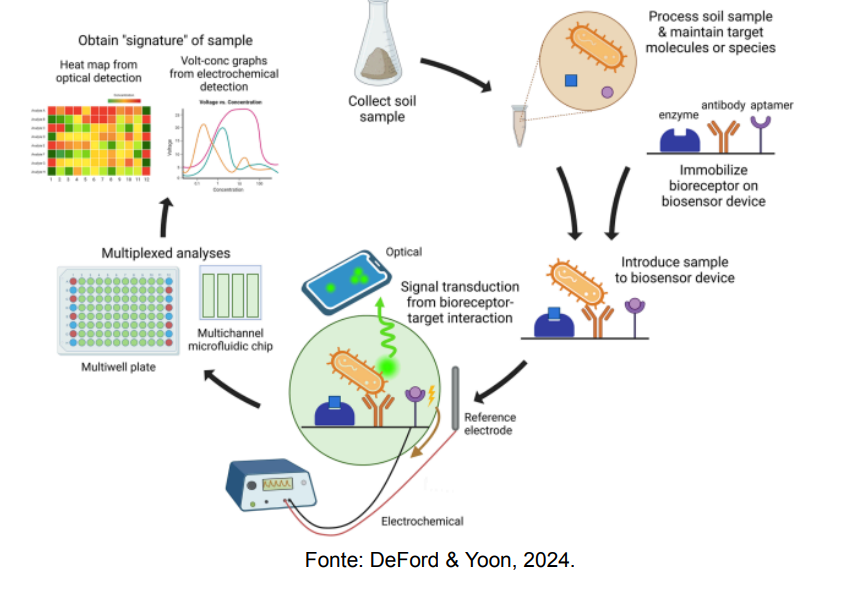
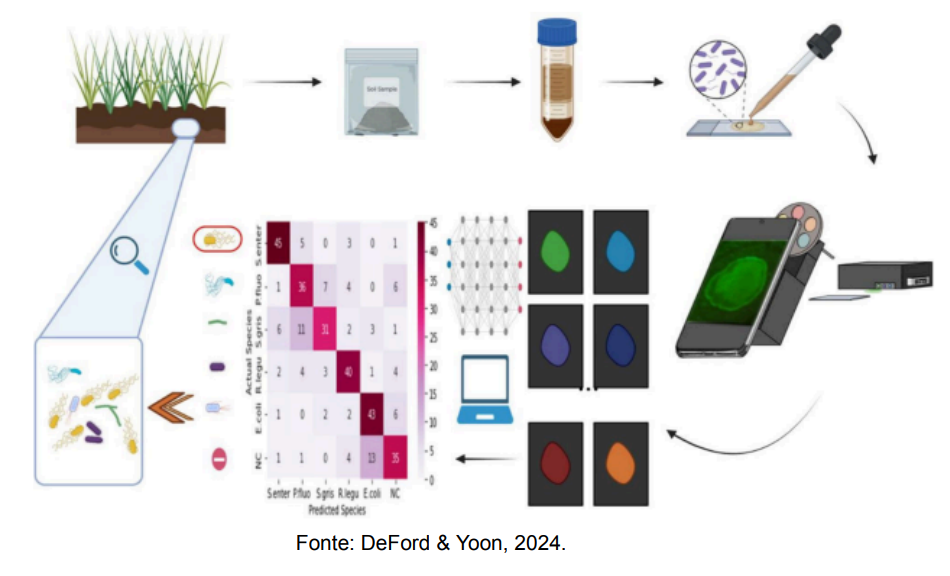
● Ferramentas de bioinformática
Há uma variedade de ferramentas para realizar análises de sequenciamento e de identificação de microrganismos. Sendo alguns mais conhecidos nessa área são:
● Kraken2 e Bracken: classificador taxonômico de sequências que atribui designações taxonômicas a sequências de DNA, examinando os k-mers dentro de uma sequência consultada e utiliza as informações contidas nesses k-mers para realizar consulta em banco de dados (o qual mapeia os k-mers para o ancestral comum mais recente de todos os genomas conhecidos por conterem um determinado k-mer). Enquanto Bracken é uma ferramenta relacionada que, adicionalmente, estima abundâncias relativas de espécies ou gêneros;
● MG-RAST: servidor open source para anotação e análises comparativas metagenômicas. O servidor fornece muitos métodos para acessar diferentes tipos de dados, incluindo reconstruções filogenética e metabólica, e possui a habilidade de comparar o metabolismo e as anotações de mais de um organismo;
● SmashCommunity: pipeline de anotação e análise de metagenômica independente, com adequação de dados de tecnologias de sequenciamento de Sanger e 454. Pode ser estendido para suportar software adicional e ter tarefas metagenômicas essenciais como montagem e previsão de genes. Estima a filogenética quantitativa e as composições funcionais de metagenoma, comparando composições de múltiplos metagenomas e produz representações visuais intuitivas de outras análises;
● Mothur: pacote de software amplo que permite aos usuários utilizar apenas um software para ter análise de dados de sequência da comunidade. Oferece a habilidade de fornecer de sequências brutas para a geração de ferramentas de visualização a fim de descrever a diversidade α e β.
● Serviços Protos
Quando é adquirido uma amostra de solo e quer analisar a biodiversidade
microbiana nessa amostra, é complicado ter isolamento de cada espécie e ainda especificar
cada uma, além de ficar claro como é a dinâmica microbiana do ambiente (como a
competição, mutualismo, amensalismo e entre outros tipos de relações interespécies).
Assim, a Protos trabalhando na área da Bioinformática e na prática laboratorial pode atuar
em:
● Montagem de genomas por meio de sequenciamentos em fragmentos curtos e reconstrução de genomas e transcritos;
● Similaridade entre as sequências de DNA e proteínas, com o software BLAST e seus derivados;
● Predizer genes, promotores e genes de RNAs (como tRNA, rRNA e até mesmos os não codificadores de proteínas) pela análise de sequências de DNA obtendo a criação de mapas de anotação funcional;
● Análise filogenética das espécies utilizando softwares mais avançados e expertise técnica;Auxiliar no planejamento de práticas laboratoriais e integração com práticas bioinformatas;
● Deixar as interpretações dos resultados de maneira mais clara e que seja mais facilitado para aplicar.

Referências:
Análise de Bioinformática. Protos Biotec Jr., São Paulo, 2024. Disponível em: https://protosbiotec.com/areasdeatuacao/analisebioinformatica. Acesso em: 5 nov. 2025.
ARUMUGAM, Manimozhiyan et al. SmashCommunity: a metagenomic annotation and analysis tool. Bioinformatics, v. 26, n. 23, p. 2977–2978, 1 dez. 2010. DOI: 10.1093/bioinformatics/btq537. Disponível em: https://www.bork.embl.de/software/smash/index.html. Acesso em: 5 nov. 2025.
BANSAL, Arvind K. Bioinformatics in microbial biotechnology – a mini review. Microbial Cell Factories, v. 4, n. 19, 2005. DOI: 10.1186/1475-2859-4-19. Disponível em: https://pmc.ncbi.nlm.nih.gov/articles/PMC1182391/pdf/1475-2859-4-19.pdf. Acesso em: 5 nov. 2025.
BHATT, Pankaj; BARH, Anupam. Bioinformatic Tools to Study the Soil Microorganisms: An In Silico Approach for Sustainable Agriculture. In: CHOUDHARY, D. K. et al. (ed.). In Silico Approach for Sustainable Agriculture. Singapore: Springer Nature, 2018. p. 169-182. DOI: 10.1007/978-981-13-0347-0_10.
CENTER FOR QUANTITATIVE LIFE SCIENCES. Kraken 2 Manual. Corvallis, OR: Oregon State University, [s. d.]. Disponível em: https://software-cqls-oregonstate-edu.translate.goog/updates/docs/kraken2/MANUA L.html. Acesso em: 5 nov. 2025.
DEFORD, Lexi; YOON, Jeong-Yeol. Soil microbiome characterization and its future directions with biosensing. Journal of Biological Engineering, v. 18, n. 50, p. 1-17, 2024. DOI: 10.1186/s13036-024-00444-1.
EDWIN, Niranjana Rose et al. An in-depth evaluation of metagenomic classifiers for soil microbiomes. Environmental Microbiome, v. 19, n. 19, p. 1-17, 2024. DOI: 10.1186/s40793-024-00561-w.
EMBRAPA. Sequenciamento de microrganismos do solo pode agir sobre doenças, produtividade e sustentabilidade agrícola. Embrapa, Brasília, DF, 9 maio 2025. Notícias. Disponível em: https://www.embrapa.br/busca-de-noticias/-/noticia/100395254/sequenciamento-de
microrganismos-do-solo-pode-agir-sobre-doencas-produtividade-e-sustentabilidade agricola. Acesso em: 5 nov. 2025.
FORMIGHIERI, Eduardo Fernandes. A bioinformática na pesquisa de microrganismos. Agroenergia em Revista, Brasília, DF, n. 5, p. 42-43, dez. 2012. Disponível em: https://www.infoteca.cnptia.embrapa.br/bitstream/doc/955049/1/Agroenergiaemrevist an.5EduFormigh.pdf. Acesso em: 5 nov. 2025.
GARG, Diksha et al. Cutting edge tools in the field of soil microbiology. Current Research in Microbial Sciences, v. 6, p. 1-18, 2024. DOI: 10.1016/j.crmicr.2024.100226.
LANGMEAD, Ben. Kraken 2/Bracken/Centrifuge AWS indexes. [S. l.]: GitHub Pages, [s. d.]. Disponível em: https://benlangmead.github.io/aws-indexes/k2. Acesso em: 5 nov. 2025.
LUCACIU, Rares et al. A Bioinformatics Guide to Plant Microbiome Analysis. Frontiers in Plant Science, v. 10, n. 1313, p. 1-18, out. 2019. DOI: 10.3389/fpls.2019.01313.
MG-RAST Help – FAQ. [S. l.]: Argonne National Laboratory, [s. d.]. Disponível em: https://help.mg-rast.org/faq.html. Acesso em: 5 nov. 2025.
SCHLOSS, Patrick D. et al. Introducing mothur: Open-Source, Platform-Independent, Community-Supported Software for Describing and Comparing Microbial Communities. Applied and Environmental Microbiology, v. 75, n. 23, p. 7537–7541, dez. 2009. DOI: 10.1128/aem.01541-09. Disponível em: https://pmc.ncbi.nlm.nih.gov/articles/PMC2786419/pdf/1541-09.pdf. Acesso em: 5 nov. 2025.
TEIXEIRA, P. Amostra metagenômica: como ela revela a vida microbiana do solo? Protos Biotec Jr., São Paulo, 2025. Blog. Disponível em: https://protosbiotec.com/blog/amostra-metagenomica-como-ela-revela-a-vida-microb iana-do-solo. Acesso em: 5 nov. 2025.
