Não é novidade que a programação tem se tornado uma esfera dominante das áreas do conhecimento. Conhecendo as linguagens de programação como quem é fluente em um idioma ou dialeto, os profissionais técnicos ou especialistas nas ciências da computação abstraem a realidade de maneira minuciosa, a interpretam em formato de código e delegam funcionalidades específicas para uma máquina, com a finalidade de realizar uma tarefa relacionada com a sua abstração da realidade. A função mais básica da computação é justamente essa: a resolução de problemas. E com isso, surgiu o Biopython.
No entanto, não é de qualquer maneira que uma máquina pode entender as tarefas delegadas pelo programador. Para isso, é necessária uma linguagem de programação, como o Biopython! Não é comum associarmos a biologia à programação, mas os sistemas biológicos e bioquímicos podem ser – dadas as devidas proporções – tratados como sistemas determinísticos, ou seja, que produzem resultados baseados somente em condições iniciais, e isso é o bastante para que modelos computacionais e matemáticos possam ser criados.
Biopython é uma biblioteca, um conjunto de códigos e funções já escritos que podem ser aplicados a um programa em Python a fim de análises de bioinformática, como sequências genéticas, populações, filogenias, etc. Veremos um pouco mais sobre esse tema adiante!
O que é Python?
Sabemos que é necessária uma linguagem de programação para que o programador se comunique com a máquina. Também sabemos que Python é, dentre muitas outras, uma linguagem de programação. O que nos resta entender é por que Python é a escolha do método desta postagem e por que é uma linguagem tão popular e amplamente utilizada.
Python é uma linguagem de programação criada por Guido van Rossum em 1991. É de alto nível, interpretada, de script, funcional, imperativa e orientada a objetos. Mas o que significam esses termos?
Vejamos:
- Linguagem de alto nível: mais parecida com a linguagem humana, mais distante da maneira como a máquina executa suas funções;
- Interpretada: antes de ser lida pela máquina, é analisada por um software intérprete, que transforma o código em uma leitura possível para o computador;
- De script: significa que a leitura do código se dá de maneira cronológica. Por exemplo, é lida de cima para baixo, os códigos em linhas anteriores são obrigatoriamente interpretados antes de outros posteriores;
- Funcional: utiliza “funções” para operar os comandos;
- Imperativa: trabalha com variáveis, dá valores a essas variáveis e as manipula para que a máquina siga uma determinada ordem de comandos;
- Orientada a objetos: focada em abstrair, criar e manipular “objetos”, que são uma classe específica de elementos do código – cuja definição é tão complexa que constantemente novos artigos científicos são publicados debatendo a própria veracidade dessa classificação.
O que todos esses pontos têm em comum é a aproximação da sintaxe dos idiomas humanos. Sabendo o básico sobre as características de Python, é possível perceber que o foco da linguagem é criar um ambiente que facilite o trabalho do programador, através de códigos mais legíveis e próximos da linguagem humana, ao invés de priorizar a velocidade da execução. Veremos mais adiante por que isso é importante ao analisar o Biopython.
Como é possível integrar a programação e a biologia?
Uma das áreas mais recentes e mais estudadas das ciências naturais são as Ômicas. “Ômicas” é um termo que engloba áreas como a genômica, transcriptômica, proteômica, metabolômica, e outras derivadas. Esses campos de pesquisa lidam com a maneira como o código genético é interpretado pelos organismos vivos. Só por essa última frase é possível entender a que ponto vamos chegar!
A genômica estuda a sequência do genoma diretamente e como a ordem em que cada molécula de DNA é arranjada influencia a manifestação de características no organismo como um todo. O seu objeto de estudo são os nucleotídeos presentes no DNA. A imagem abaixo mostra uma molécula de DNA com seus nucleotídeos, representados pelas letras T, A, G e C.
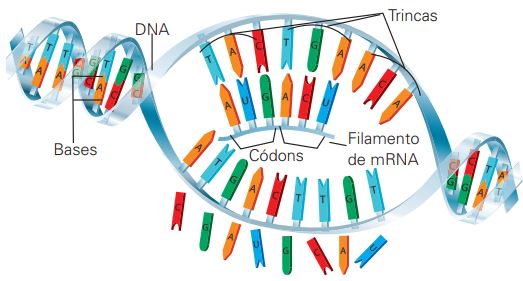
A transcriptômica estuda como todos esses nucleotídeos são lidos e copiados em uma outra molécula, chamada RNA mensageiro, também presente na imagem acima. A proteômica aborda as instruções presentes no RNA mensageiro, que virão a ser interpretadas e transformadas em proteínas ainda nas células, e estuda a estrutura dessas proteínas. Por fim, a metabolômica integra toda a produção de proteínas e outros compostos no organismo e estuda como eles interagem entre si, através de vias metabólicas, e como essas vias desencadeiam em funções básicas do organismo.
Fonte: https://www.coladaweb.com/biologia/biologia-celular/respiracao-celular
Com essa última seção do texto, uma breve explicação biológica, ficou um pouco mais fácil de imaginar como a programação pode se integrar com a biologia, em especial, com a bioquímica e a biologia molecular. Em todas as vezes que as palavras “código”, “ler”, “interpretar”, “transformar”, “ordem”, “sequência”, “função” e “modelo” foram mencionadas, o leitor deve ter pensado que esses processos abrem portas para que alguma linguagem de programação crie maneiras análogas de simulá-los. E está mais do que certo!
Nessa situação, entra o Biopython, uma maneira de modelar as informações biológicas em dados virtuais, simular os processos de transformação desses dados, e sintetizá-los de volta em informações e compostos biológicos.
Introdução ao Biopython
Como já foi comentado anteriormente, Biopython é uma biblioteca de código aberto que pode ser aplicada a um código em Python a fim de realizar algumas análises biológicas. A biblioteca é o resultado de um projeto idealizado por Jeff Chang e Andrew Dalke, com a finalidade de criar um armazém de métodos disponíveis a todos que venham a estudar as ciências naturais, e de maneira cumulativa e contributiva. Pela natureza do Python, toda contribuição gerada por um usuário do programa pode ser complementada por outro, e assim sucessivamente, de maneira que todos contribuem para o crescimento do projeto como um todo.
Nas palavras dos próprios criadores: “Nós acreditamos que esse campo pode avançar mais rapidamente se bibliotecas que realizam funções comuns estiverem disponíveis. Então, esperamos criar uma fonte central de ferramentas de bioinformática de alta qualidade que os pesquisadores possam utilizar”.
A necessidade fundamental da bioinformática é justamente essa maneira de adquirir informações de fontes biológicas e inseri-las em estruturas em Python. Para isso, o Biopython apresenta uma biblioteca de Scanners e Consumers, cujas funções são reconhecer informações em sequências e extrair essas informações, respectivamente.
As sequências são geralmente apresentadas em arquivos do tipo FASTA, quando lidando com genomas, e PDB, quando lidando com proteínas.
Pense na seguinte sequência:
ATGAAAGCAATTTTCGTACTGAAAGGTTGGTGGCGCACTTGA.
Um código básico que viria a ser implementado seria:
>>> from Bio.Seq import Seq
>>> from Bio.Alphabet import generic_dna
>>> gene = Seq(“ATGAAAGCAATTTTCGTACTG”
… “AAAGGTTGGTGGCGCACTTGA”,
… generic_dna)
>>> print gene.transcribe()
AUGAAAGCAAUUUUCGUACUGAAAGGUUGGUGGCGCACUUGA
>>> print gene.translate(table=11)
MKAIFVLKGWWRT*
Essas linhas, em ordem, acessam a biblioteca do Biopython, inserem uma certa sequência de nucleotídeos do DNA, transcrevem em uma sequência de RNA e, por fim, traduzem o RNA em uma sequência de aminoácidos! Vale ressaltar que esse procedimento não serve apenas para sequenciamento de DNA, mas também para árvores filogenéticas, dinâmicas de populações, progressões evolutivas, entre outros, ainda que a primeira seja a aplicação mais aclamada.
A partir dessa interpretação básica fornecida pelo Biopython, outros softwares podem progredir no estudo bioquímico, como o BLAST e o BLASTX, que relacionam sequências de aminoácidos às suas estruturas; AlphaFold, que prediz com altíssima precisão o enovelamento das proteínas; e PyMol, que analisa estruturas químicas no geral.
Perspectivas futuras.
Os criadores do projeto continuam a expandir cada vez mais as bibliotecas. Os seus próximos objetivos declarados são a incorporação de uma interface visual mais amigável ao usuário, uma interação maior com outros softwares, biogeografia aplicada à genética de populações, além de uma possível Biopython 2. Este amplo projeto que partiu de apenas duas pessoas continua a contribuir ativamente para os estudos de vanguarda das ciências biológicas.
Deseja conferir mais conteúdos sobre o mundo da bioinformática e suas análises? Clique aqui e confira já em nosso blog!

Referências
BERMAN, H. M. The Protein Data Bank. Nucleic Acids Research, v. 28, n. 1, p. 235–242, 1 jan. 2000.
BIOPYTHON ORG. Biopython, 2021. Página Inicial. Disponível em: <https://biopython.org>. Acesso em: 23 set. 2022.
CHAPMAN, B.; CHANG, J. Biopython: Python tools for computational biology. ACM SIGBIO Newsletter, v. 20, n. 2, p. 15–19, ago. 2000.
COCK, P. J. A. et al. Biopython: freely available Python tools for computational molecular biology and bioinformatics. Bioinformatics, v. 25, n. 11, p. 1422–1423, 1 jun. 2009.
NELSON, D.L.; COX, M.M. Princípios de Bioquímica de Lehninger. 6. ed. São Paulo: Artmed, 2014. p. 501 – 542.
SANNER, M. F. et al. INTEGRATING COMPUTATION AND VISUALIZATION FOR BIOMOLECULAR ANALYSIS: AN EXAMPLE USING PYTHON AND AVS. Biocomputing ’99. Anais…Mauna Lani, Hawaii, USA: WORLD SCIENTIFIC, dez. 1998. Disponível em: <http://www.worldscientific.com/doi/abs/10.1142/9789814447300_0039>. Acesso em: 26 set. 2022.
